El pasado 15 de diciembre de 2014 se celebró la jornada para la presentación de resultados del proyecto FI4VDI [1, 2], en el Complejo Asistencial Universitario de León. El proyecto propone una infraestructura informática en la nube utilizando los recursos disponibles de distintos centros de cálculo intensivo situados en el territorio del Espacio SUDOE, con el fin de garantizar la protección de datos de los usuarios, la conformidad y la seguridad de la información. El lanzamiento de este servicio persigue la mejora de la competitividad y de las economías en los sectores específicos.
Para cumplir con los objetivos del proyecto se ha utilizado OpenNebula, para desplegar una plataforma que permita a sus usuarios el acceso a recursos físicos heterogéneos bajo demanda, provenientes de los centros de supercomputación. El acceso a la plataforma es público, aunque deberá solicitarse previamente al consorcio del proyecto.
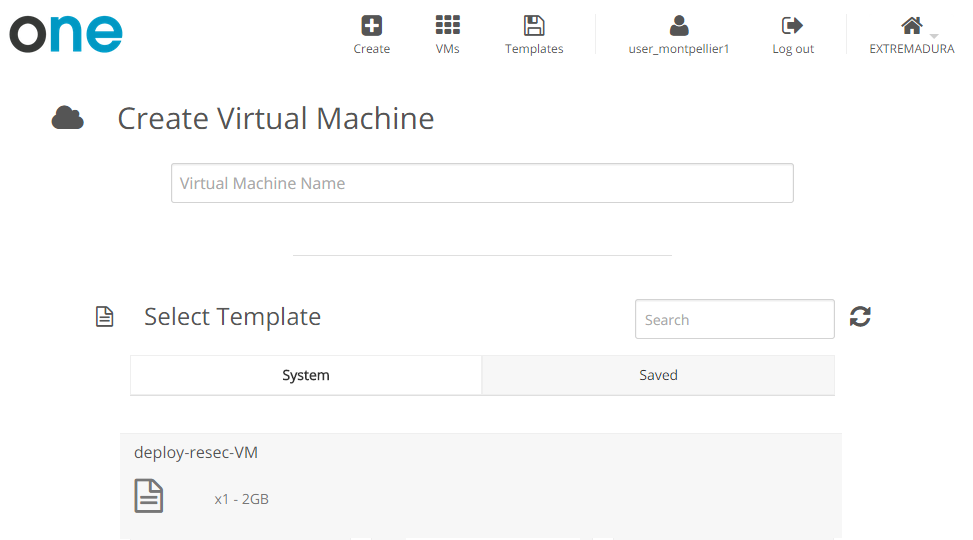
Figura con una captura de pantalla de la interfaz de OpenNebula, donde se ve cómo un usuario puede desplegar la MV con el prototipo a partir de la plantilla mostrada en la imagen:
En la jornada destacaron las ponencias donde se presentaron los prototipos desarrollados para la plataforma. Se presentaron cuatro, dos de los cuales estaban relacionados con la sanidad. Concretamente, la Fundación COMPUTAEX [3] presentó su “Entorno semi-automático de diagnóstico e investigación aplicado a la ultrasecuenciación genética en un centro de supercomputación”, presentado también en el congreso IberGrid 2014 en Aveiro [4]. Este consiste en la automatización del lanzamiento de las tareas que realizan las fases computacionales de un experimento de resecuenciación genética (alineamiento, detección y anotación de variantes). Además, se proporcionan herramientas de apoyo para que el especialista interprete, de forma manual, los resultados obtenidos.
La implementación del prototipo ha requerido el cumplimiento de dos objetivos: el primero la obtención de la información que los genetistas necesitan como apoyo al estudio de enfermedades hereditarias. El proceso que se lleva a cabo puede consultarse tanto en el artículo para IberGrid como en la memoria del sub-proyecto Estirpex [5], del proyecto CENITAL de COMPUTAEX.
El segundo objetivo ha sido plasmar ese proceso en una imagen de OpenNebula, de tal manera que un usuario que disponga de credenciales para acceder a la plataforma pueda desplegar una máquina virtual preparada para procesar sus secuencias genéticas. Para ello, se ha instalado CentOS 6 en una imagen persistente de OpenNebula, de tal manera que al borrar la máquina de instalación del SO los datos no se pierdan. Mediante la creación de otra máquina virtual que ahora arranque desde CentOS se han añadido los scripts e instalado el software del prototipo. Posteriormente, se ha copiado la imagen, que será la que se use en la plantilla para que los usuarios desplieguen sus propias máquinas virtuales. La diferencia es que ahora la copia no es persistente, por lo que cada vez que un usuario despliegue una máquina su imagen será privada, y cuando la borre, borrará también sus datos.
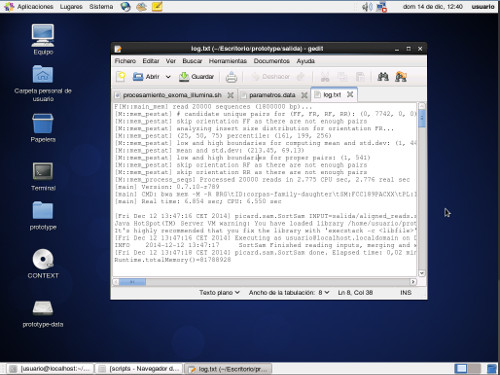
Figura con el contenido del log de salida del software utilizado para procesar las secuencias genéticas:
Es destacable mencionar que todos los ficheros de bases de datos de variaciones están alojados en una máquina adicional, exportada mediante NFS. De esta forma, la imagen de los usuarios es muy ligera, y el despliegue muy rápido. Además, OpenNebula permite que sus usuarios puedan modificar las prestaciones de las máquinas desplegadas.
El prototipo ha sido desarrollado en colaboración con el Servicio de Inmunología y Genética Molecular del Hospital San Pedro de Alcántara de Cáceres [6].
- FI4VDI http://fi4vdi-sudoe.org/index.php/es/
- Encuentro FI4VDI https://encuentrofi4vdi.fcsc.es/
- COMPUTAEX http://www.computaex.es/
- Presentación paper IberGrid 2014 http://www.computaex.es/noticias/05092014-computaex-presenta-articulo-secuenciacion-genetica-ibergrid-2014
- M.A. Rourich, J.L. González-Sánchez, P.G. Rodríguez, F. Lemus: «Automatic Genetic Sequences Processing Pipeline in the Cloud for the Study of Hereditary Diseases», Congreso Ibergrid (8th Iberian Grid Infrastructure Conference), pp.129-142, Aveiro (Portugal), September 8-10, 2014
- Estirpex http://www.computaex.es/enlaces/publicaciones/estirpex
- Servicio de Inmunología y Genética Molecular del Hospital San Pedro de Alcántara de Cáceres http://www.areasaludcaceres.es/modulos/mod_organigrama/pub/ficha_servicios.php?id=4
Entradas relacionadas con este post en el blog: